HelixFold
HELIX-Fold 端到端地学习蛋白质结构,通过协同学习蛋白质的多序列比对(MSA)和氨基酸对(pairwise)的表征,将蛋白质序列的进化信息、蛋白质结构的物理和几何约束信息结合到深度学习网络中。利用大规模无标注数据强化蛋白质表示能力,构建的全国产化软硬件适配的高效蛋白质结构分析大模型。
用图形桌面模式如何提交任务
一、 提交流程
登录 Fastone 平台控制台;
数据管理上传计算文件;
在首页,点击【提交作业】-搜索【HelixFold】-点击【HelixFold】应用-选择机器配置,进行启用机器;
上传计算文件,设置资源参数;
二、单机模式
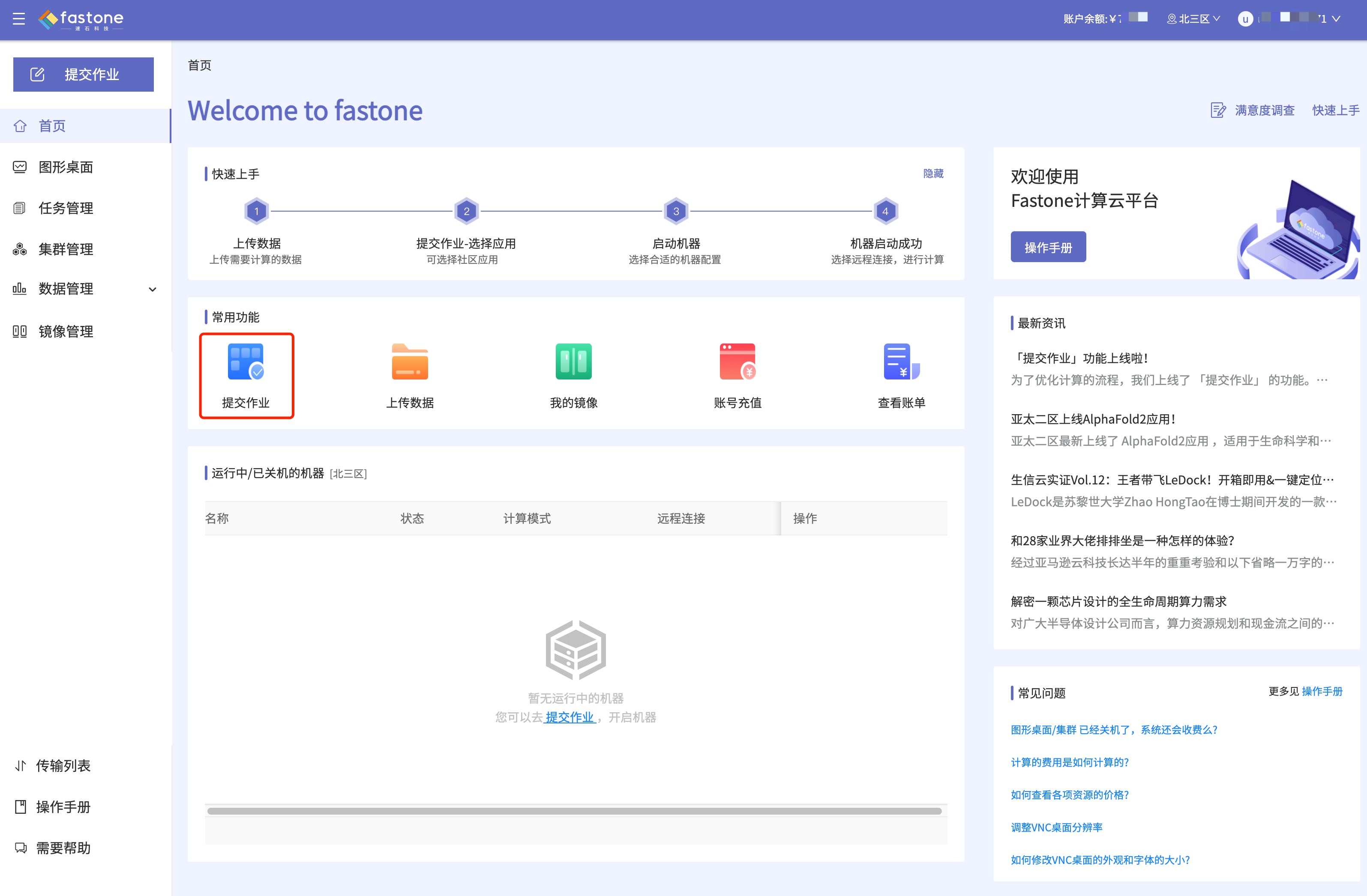
Step 1:首页选择【提交作业】,如图:
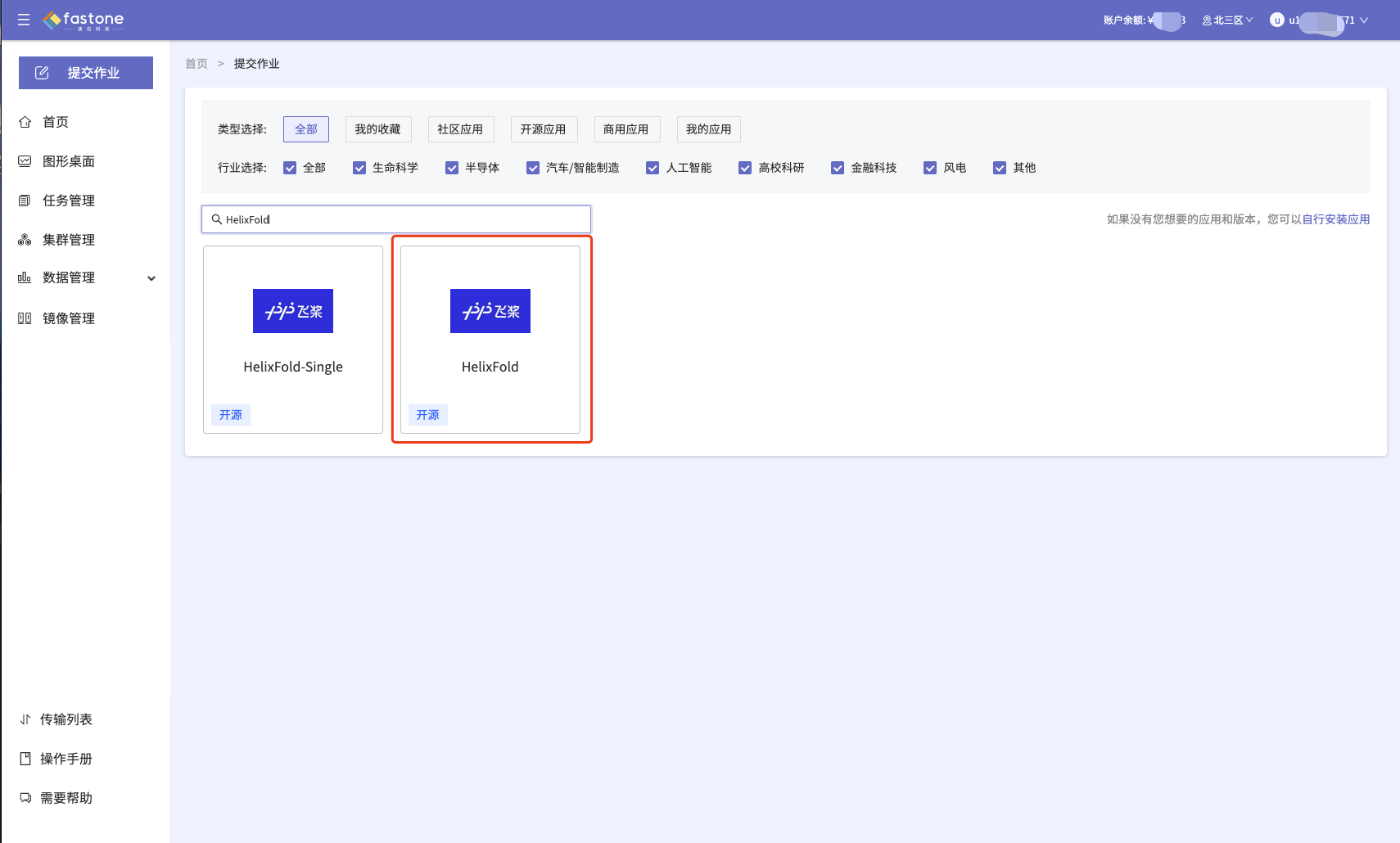
Step 2:选择【HelixFold】应用进行启用,如图:
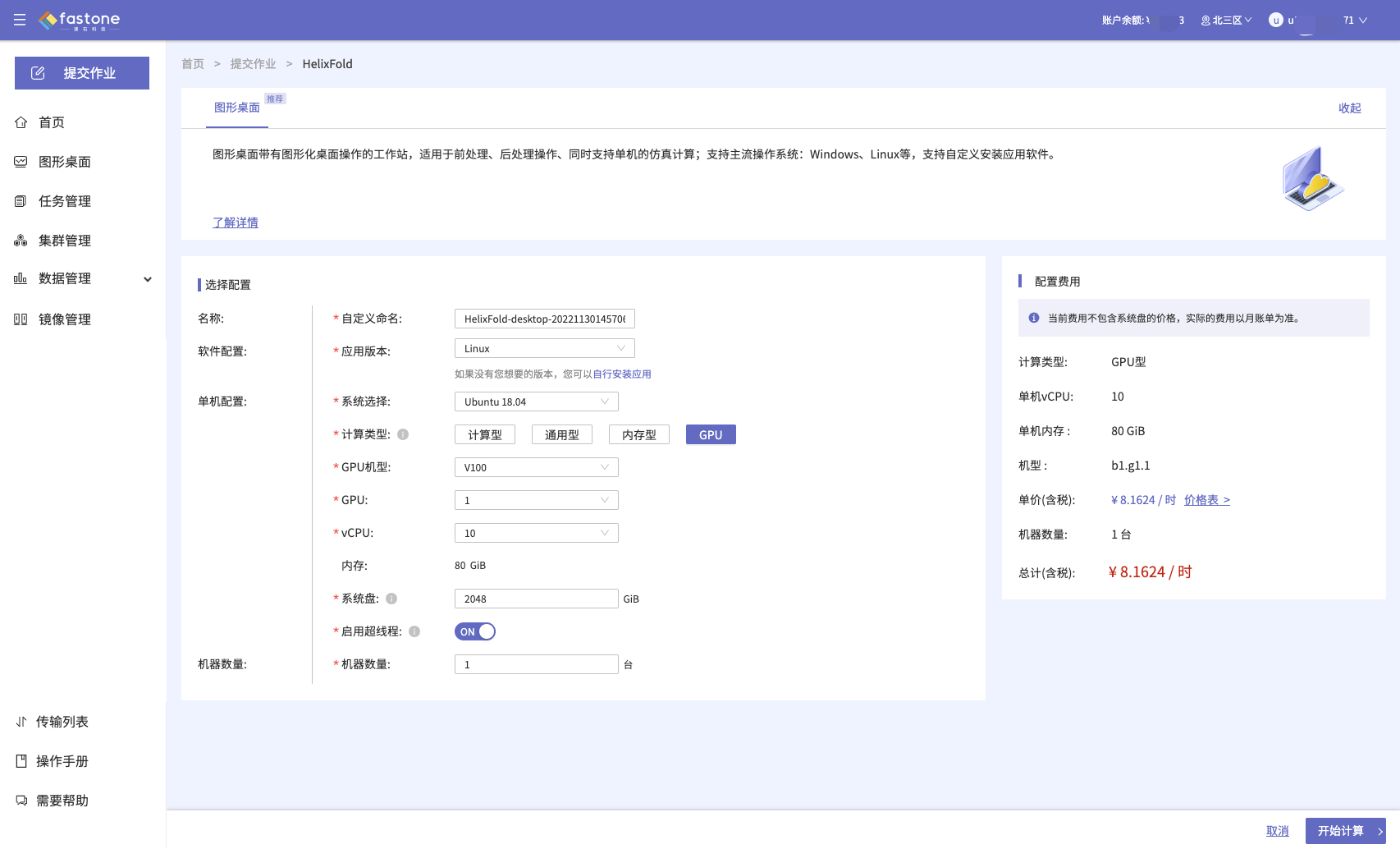
- 机器配置:系统选择为Ubuntu18.04,选择计算类型为GPU,系统盘为2048GiB
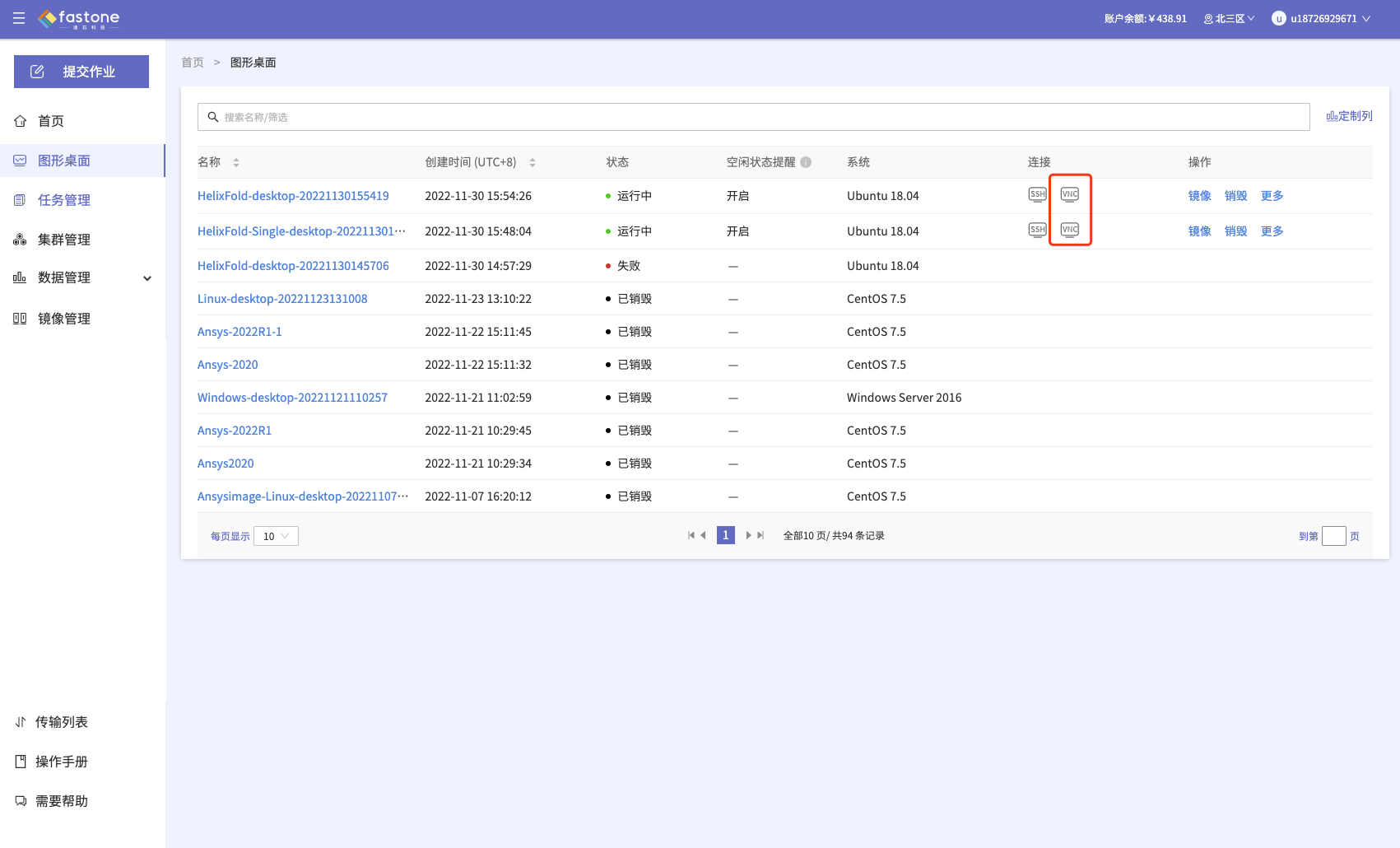
Step3: 等待机器启动,点击【图形桌面】,点击VNC连接。
Step 4:进入图形界面,打开Terminal,如图:

Step 5:运行命令
- helixfold应用在/helixfold目录下
Bash
source /helixfold/conda_env.sh
conda activate helixfold
bash run_helixfold.sh <input_file> <output_path>
1
2
3
4
2
3
4
- input_file:为输入的蛋白质长度
- output_path: 结果输出目录
Step 6:计算结果
等待一段时间后,在输出目录会有结果文件,则运行完成。
