用集群模式如何提交任务
一、 提交流程
登录 Fastone 平台控制台;
数据管理上传计算文件;
在首页,点击【提交作业】-点击【QVina】应用-选择任务模式-选择机器配置,进行启用机器;
上传计算文件,设置资源参数;
二、 操作步骤
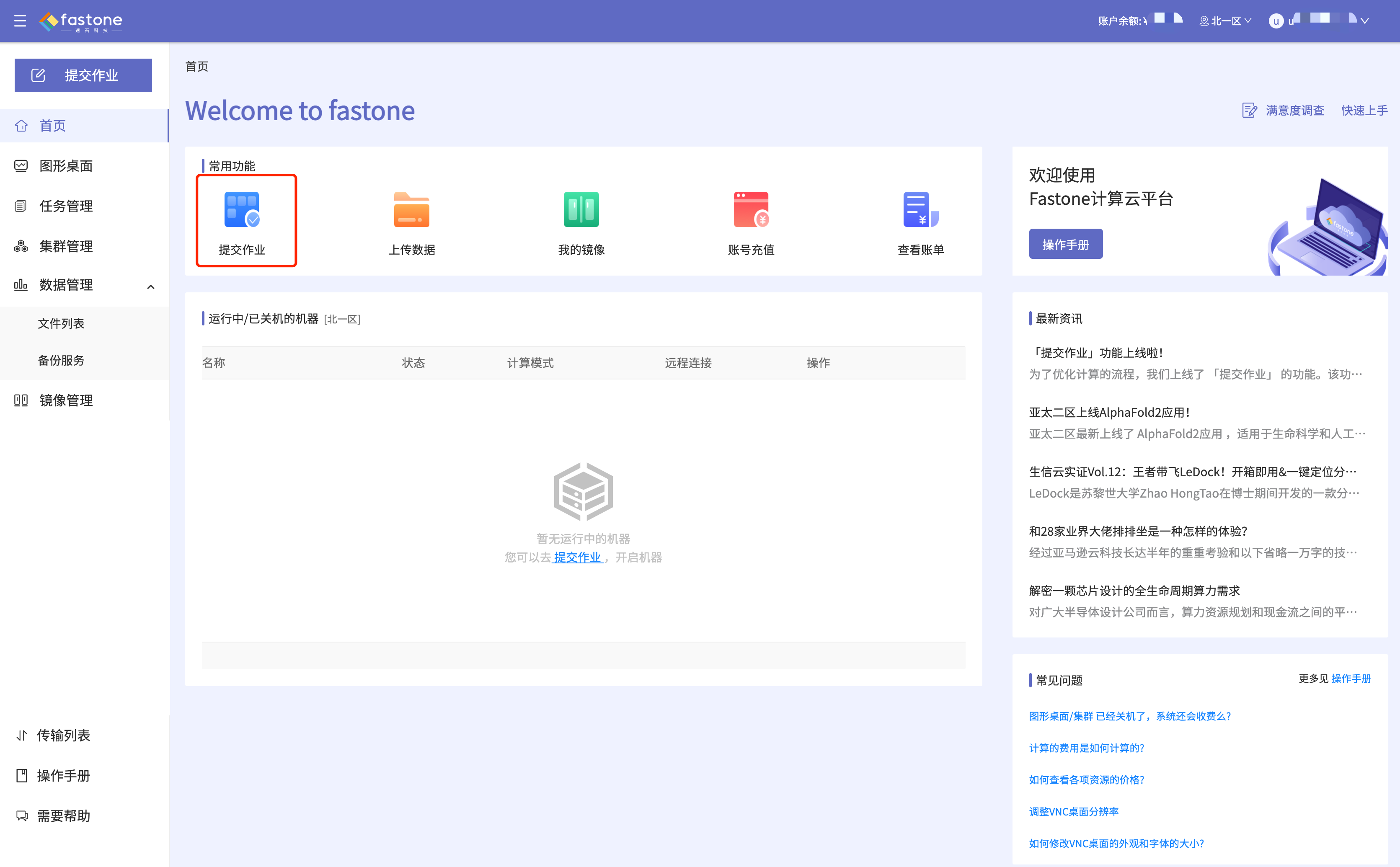
Step 1:首页选择【提交作业】,如图:
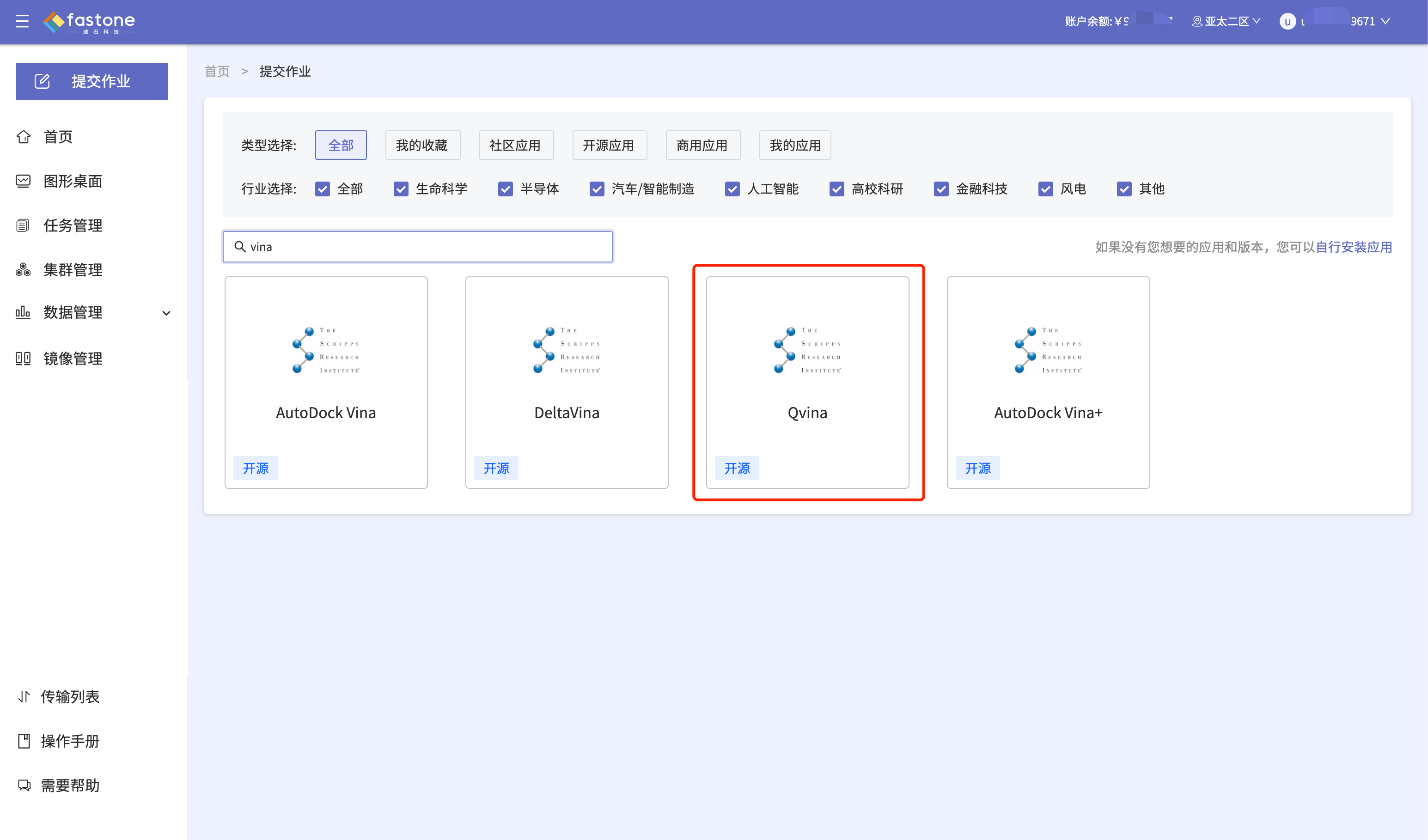
Step 2:选择应用【Qvina】
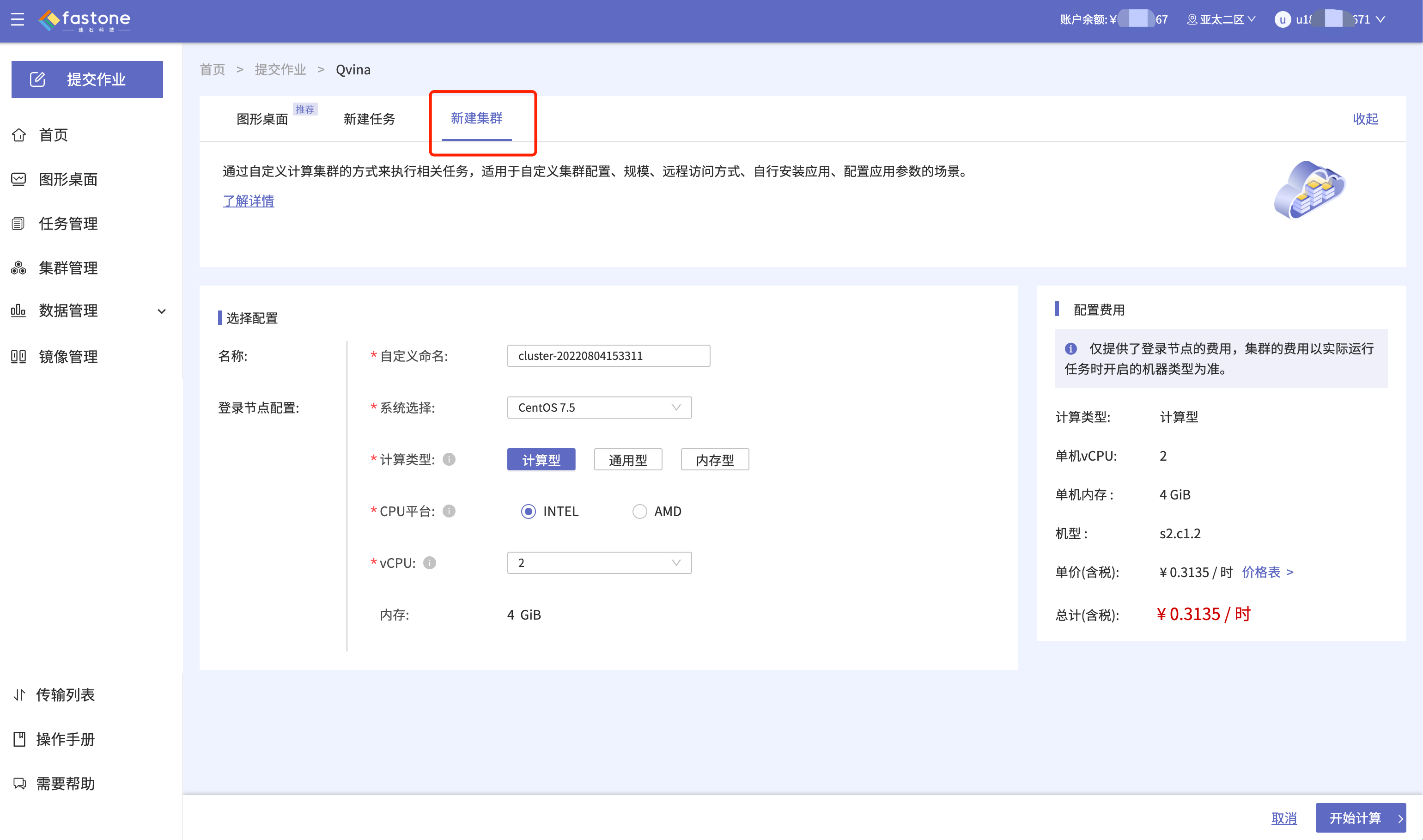
Step 3:配置集群登录节点资源配置,点击【开始计算】
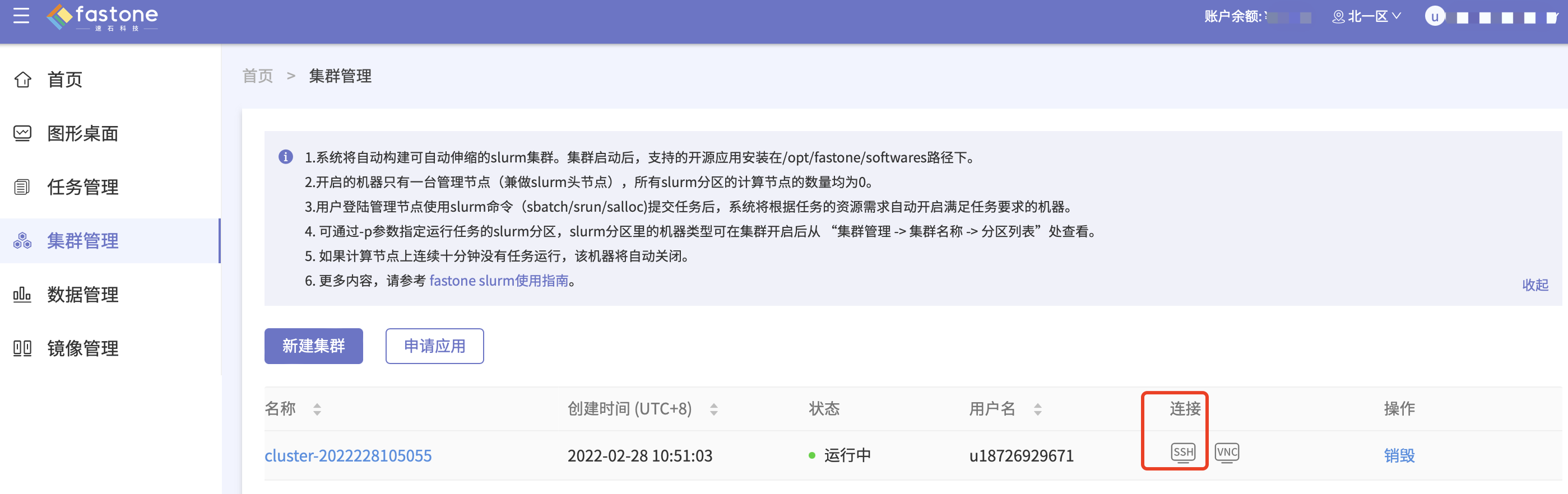
Step 4:集群管理,选择创建的集群,在连接选择【SSH】连接,如图:
三、提交任务
Step 1:通过SSH连接创建并连接集群登录节点。
Step 2. 通过数据管理上传所需的输入文件(比如上传目录~/project/vina)
Step 3. 在上传的项目文件目录下(比如~/project/vina)执行qvina流程脚本run-vina.sh
cd ~/project/vina
srun -n<NPROC> -N<NODES> /opt/fastone/softwares/virtual-screening/run-vina.sh -receptor <受体分子文件.pdb> -mol <配体分子库文件.sdf> -resname <用于定位的分子链标识如MOV.303>
1
2
2
qvina流程脚本run-vina.sh使用说明:
NPROC:并行核数;NODES:启动节点数量
vina必选参数
-receptor #受体分子文件,文件格式.pdb
-mol #配体分子库文件,文件格式.sdf
-resname #分子链标识,用于定位的分子链,自动计算中心坐标,多个分子用空格间隔(ResName.Id, eg MOV.303 GDP.302)
1
2
3
2
3
- 其它可选参数,对应conf.txt所需的参数,不设置则使用缺省值
-size_x
-size_y
-size_z
-num_modes
-energy_range
-weight_hydrogen
-affinity_filter
1
2
3
4
5
6
7
2
3
4
5
6
7
使用qvina流程脚本run-vina.sh会自动计算分子中心点(center_x、center_y、center_z),移除所选分子,启动分子对接流程,输出结果包含:含中心坐标的config文件、移除分子后的pdb文件、打分结果总表、Affinity筛选结果、分子对接结果文件分类压缩。 也可以直接使用传统qvina命令(/opt/fastone/softwares/qvina/qvina02)方式运行vina。
