NAMD
NAMD (NAnoscale Molecular Dynamics) 是一个产品级分子动力学应用程序,专为实现大型生物分子系统的高性能模拟而设计,支持Charmm、Namd和Amber等多种力场。
用新建任务模式如何提交任务
一、 提交流程
登录 Fastone 平台控制台;
数据管理上传计算文件;
在首页,点击【提交作业】-点击【NAMD】应用-选择任务模式-选择机器配置,进行启用机器;
上传计算文件,设置资源参数;
二、单机模式
Step 1:首页选择【提交作业】,如图:
Step 2:选择应用【NAMD】;
Step 3. 上传输入文件,配置运行参数;
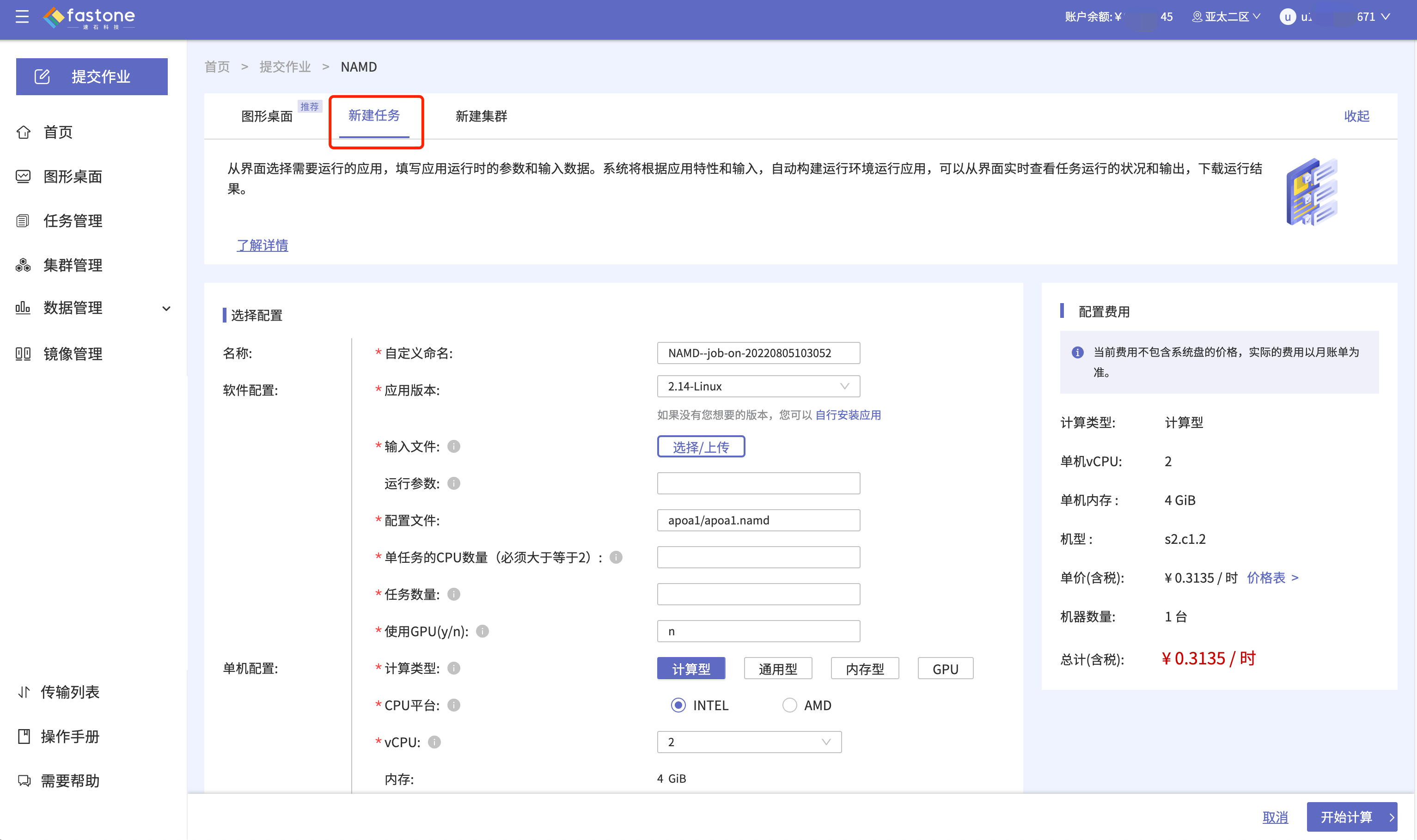
- 可以设置提交的作业名。
- 可点击感叹号查看待上传输入文件的格式、软件运行参数的注意事项等。
以ApoA1(载脂蛋白A1)的分子动力学模拟为例: 以ApoA1(载脂蛋白A1)的分子动力学模拟为例:
- 上传输入文件apoa1.tar.gz至数据管理;
输入文件中:
.namd NAMD的运行配置文件
.pdb 包含分子系统最初结构的文件
.psf PSF格式的分子结构文件
.xplor 力场参数文件
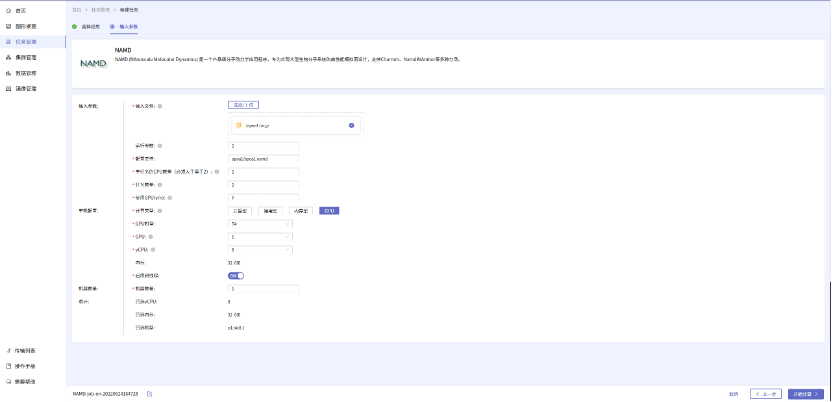
- 新建任务,输入文件选择apoa1.tar.gz,任务数量建议为2,使用GPU在集群配置无GPU时使用默认值n, 在资源配置有GPU并且要使用GPU时输入y;
- 开始执行相关任务。
Step 4. 选择硬件配置;
- 根据软件版本和使用需求,选择CPU/GPU配置。
- 节点数量:设置启动多少个并行计算的计算节点。
- 内存配比:设置各个计算节点内存大小为单节点核心数×内存配比。
Step 5. 查看作业内容汇总,并提交作业;
三、提交后的监控
提交任务,任务运行过程中可查看stdout.txt了解进度。
作业提交后可通过任务管理查看任务的输出和日志等内容。
可通过集群监控查看正在运行的计算节点资源使用率等信息。
