AlphaFold2
Alphafold2是一个预测蛋白质三维空间结构的深度学习模型,通过深度学习算法通过蛋白质氨基酸序列来预测蛋白质结构。
用图形桌面模式如何提交任务
一. 提交流程
登录 Fastone 平台控制台;
数据管理上传计算文件;
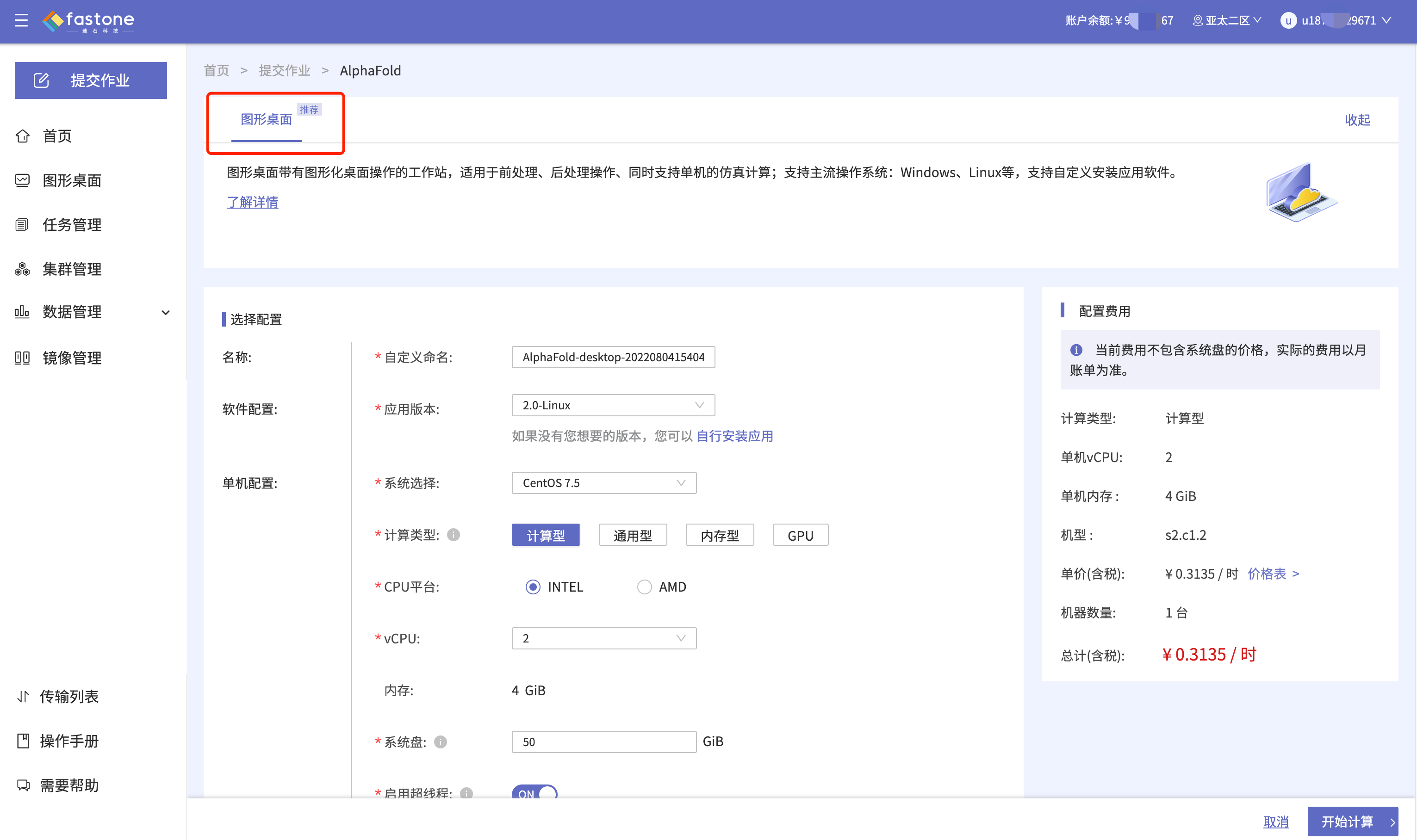
在首页,点击【提交作业】-点击【AIphaFold】应用-选择图形桌面模式-选择机器配置,进行启用机器;
上传计算文件,设置资源参数;
二、单机模式
Step 1:首页选择【提交作业】,如图:
Step 2:选择【AlphaFold2】进行启用,如图:
- GPU型机器配置:建议系统盘选择70GiB以上
- 非GPU型机器配置:建议系统盘选择70GiB以上
Step 3:在图形界面,选择刚才已创建且处于运行中的图形桌面,点击【VNC】进行远程连接 
Step 4:进入图形桌面,打开Terminal,如图:
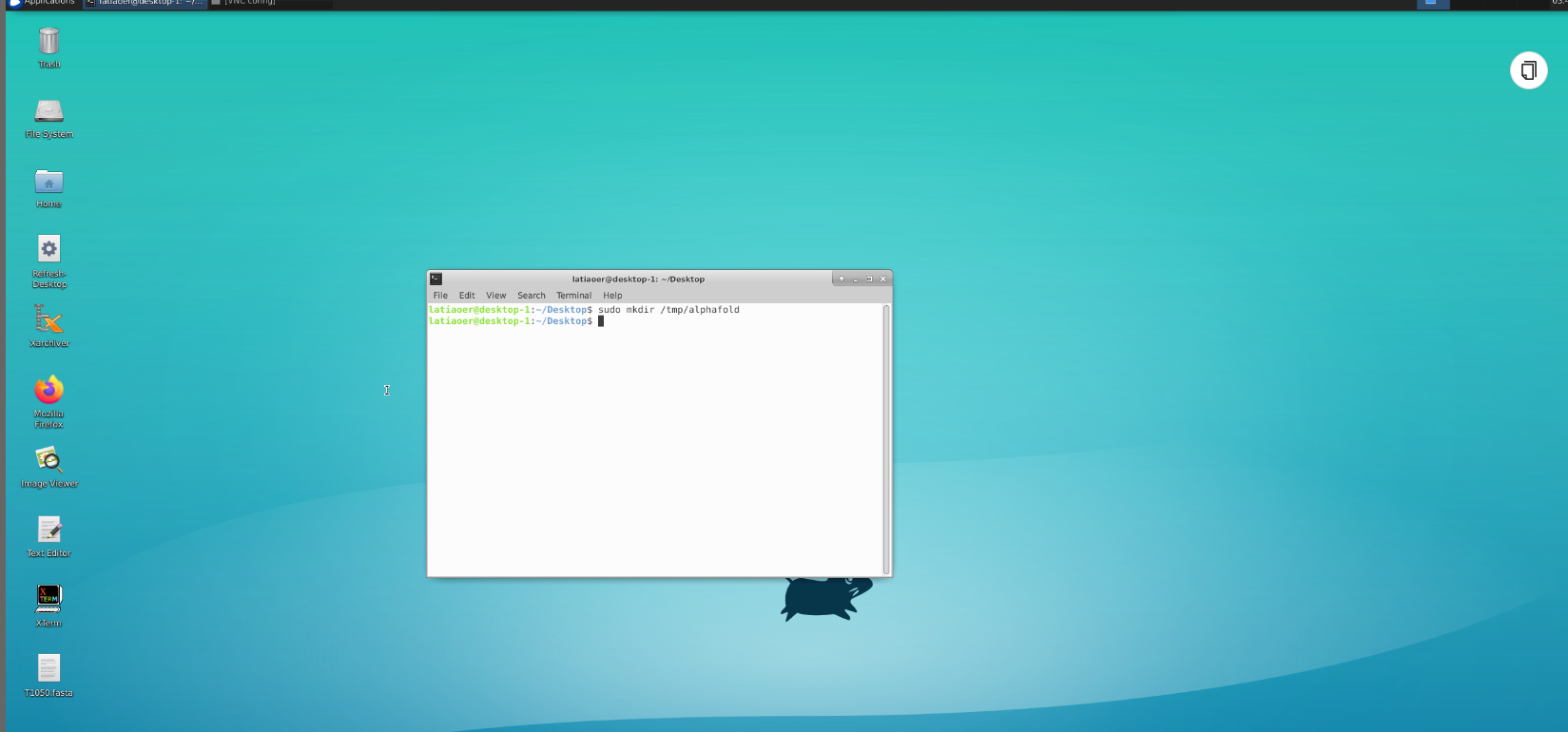
Step 5:命令行提交(例如当前用户:latiaoer)
a)、创建作业输出目录
后续作业完成后可在此目录下查看作业结果
mkdir alphafold
## alphafold的全路径为:/fastone/users/latiaoer/Desktop/alphafold
1
2
2
b)、执行命令
- GPU机型运行时间大约5.5h+
- 非GPU机型运行时间大约12h+
(选择的如果是非GPU机型,则要在如下命令中增加 --use_gpu=False)
sudo python3 /alphafold-project/alphafold-main/docker/run_docker.py --
fasta_paths=/fastone/users/latiaoer/556single.fasta --
data_dir=/opt/fastone/softwares/fastone/software/bio/alphafold --
max_template_date=2020-05-14 --
db_preset=full_dbs --
docker_image_name=alphafold:2.2.2_8 --
output_dir=/fastone/users/latiaoer/output
1
2
3
4
5
6
7
2
3
4
5
6
7
参数说明:
--fasta_paths:AlphaFold2所需要的pdb输入文件路径,可按需更改
--use_gpu :是否使用gpu机型,默认True,非gpu机型可指定 --use_gpu=False,可按需更改
--output_dir:作业执行结果输出路径,可按需更改
--db_preset: 可按需更改
reduce_dbs:针对速度和较低的硬件要求进行了优化。
full_dbs:这与 CASP14 使用的所有基因数据库一起运行。
--data_dir: 基因数据库所在目录,无需更改
--model_preset=multimer :来运行多体结构预测
Step 6:结果文件查看
进入/fastone/users/latiaoer/Desktop/alphafold

