Rosetta
Rosetta是用于大分子建模的首要套件,包括用于计算建模和分析蛋白质结构的算法,从头蛋白质设计、酶设计、配体对接以及生物大分子和大分子复合物的结构预测。
用图形桌面模式如何提交任务
一、 提交流程
登录 Fastone 平台控制台;
数据管理上传计算文件;Rosetta所需要的输入文件,需是以 .fasta 格式结尾的文件;
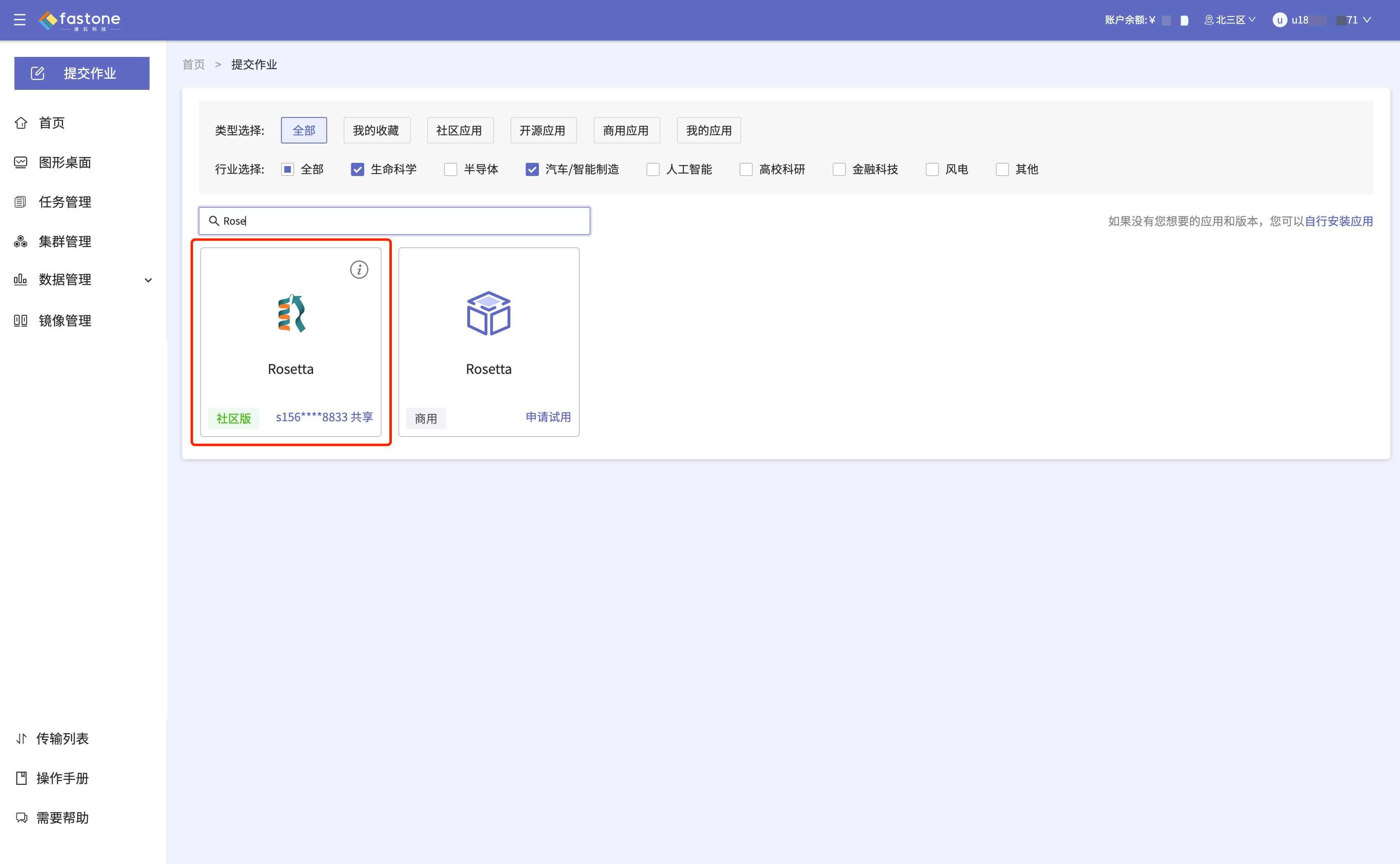
在首页,点击【提交作业】-搜索【Rosetta】-点击【Rosetta】应用-选择机器配置,进行启用机器;
上传计算文件,设置资源参数;
二、单机模式
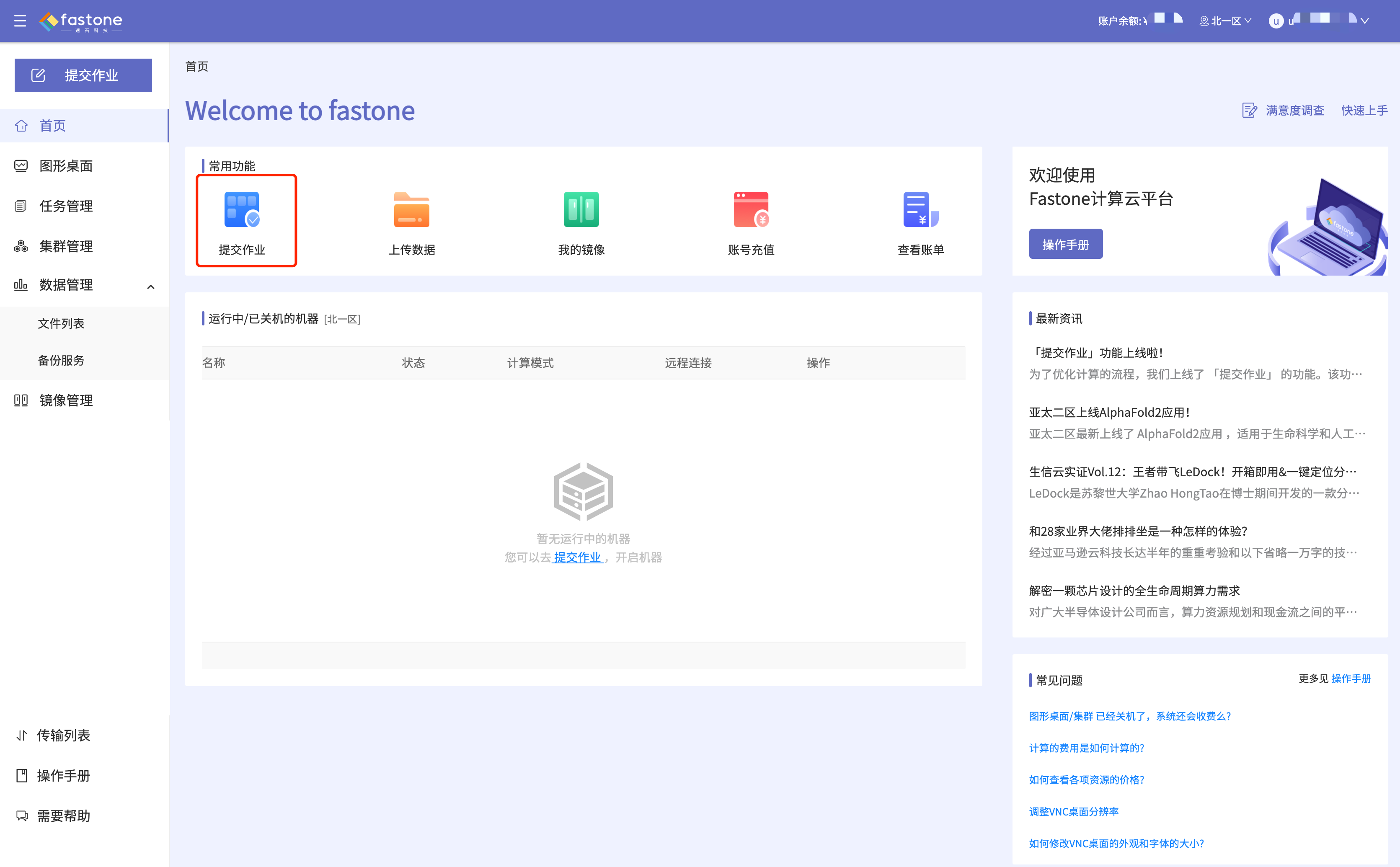
Step 1:首页选择【提交作业】,如图:
Step 2:选择【Rosetta】应用进行启用,如图:
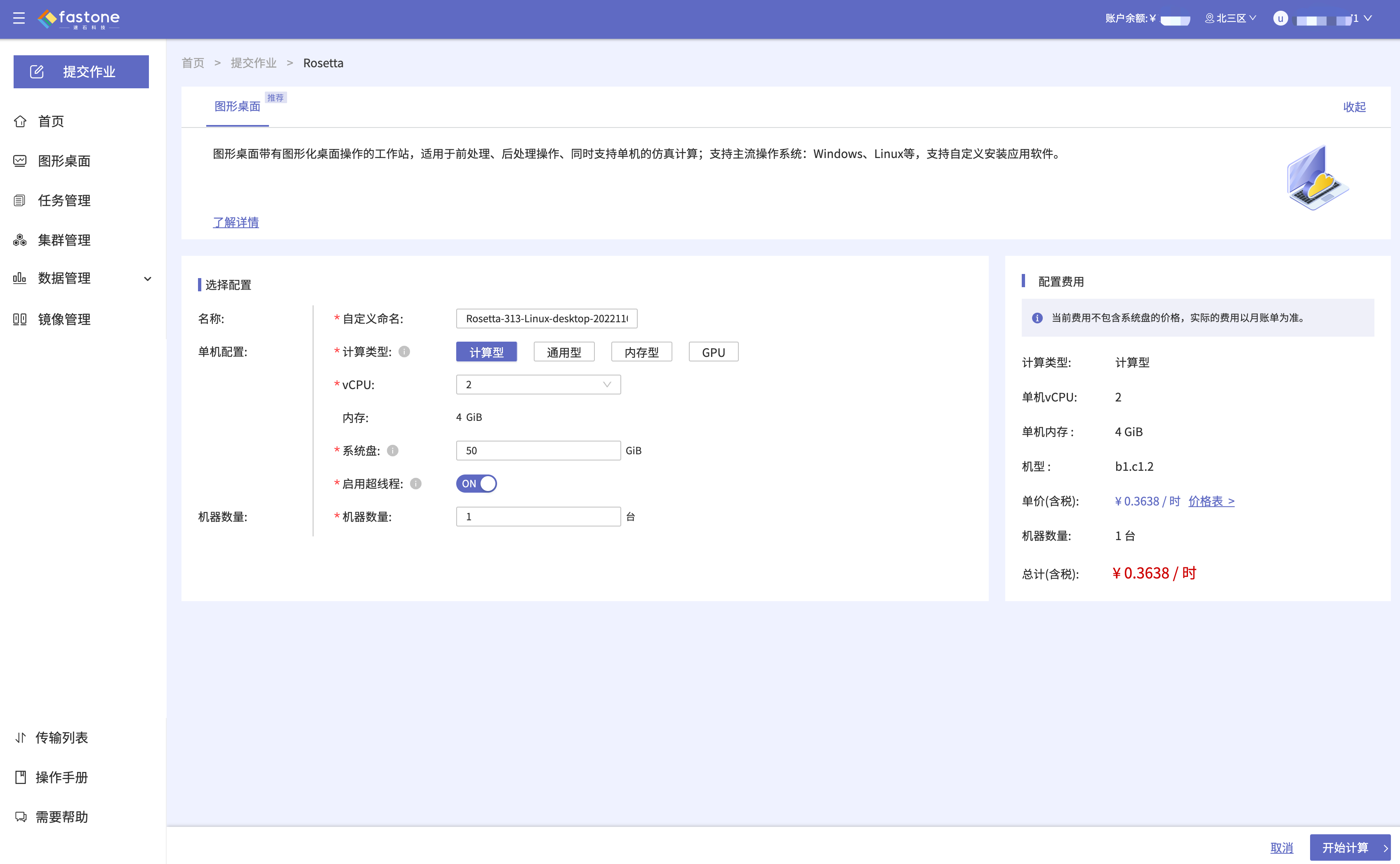
- 机器配置:建议系统盘选择50GiB以上
- 建议选择4vCPU或以上的机器进行计算,不支持2vCPU机型
- 建议系统盘选择50GiB以上
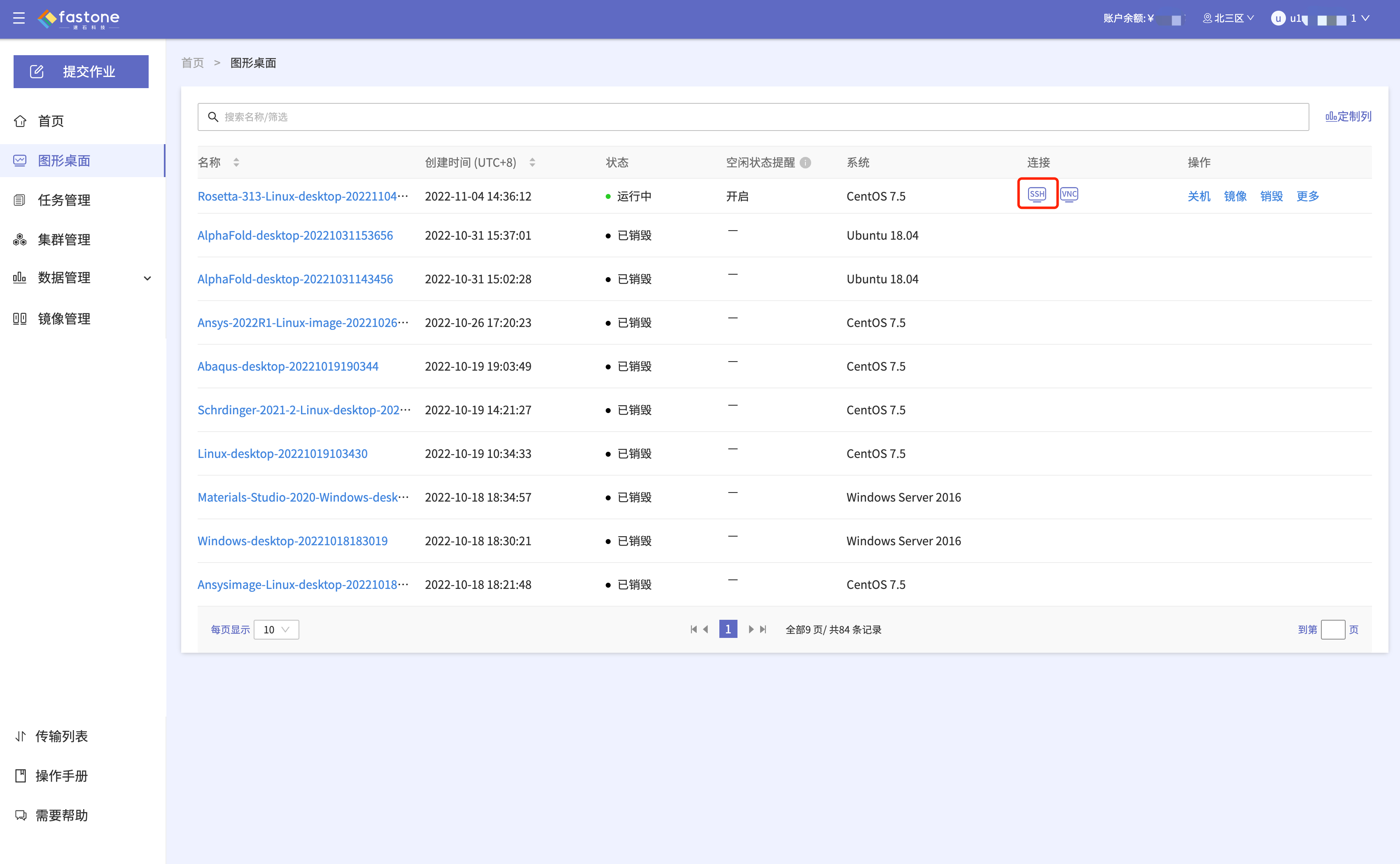
Step 3:在图形界面,选择刚才已创建且处于运行中的图形桌面,点击【SSH】进行远程连接
Step 4:进入图形界面,打开Terminal,如图:

Step 5:命令行提交
- 测试Rosetta运行状态是否正常
antibody.mpi.linuxgccrelease
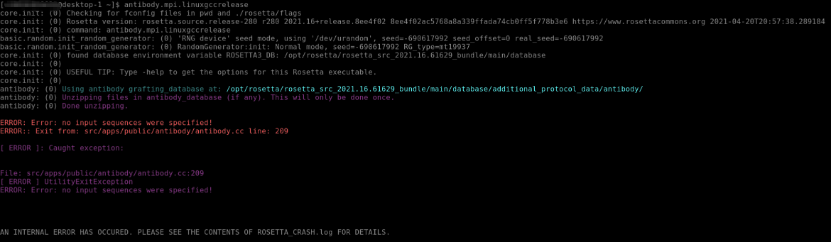
提交任务,对格式文件为fstab的蛋白质文件进行操作和提交。
Rosetta的输入文件形式比较多样,一般以PDB文件作为输入。
除此以外,不同的app要求的输入文件可能并不一样。比如在结构预测时需要定义Fragment文件、膜蛋白设计中需要使用Span文件,在Loop建模中需要定义Loop文件等。
APP目录
/opt/rosetta/rosetta_src_2021.16.61629_bundle/main/source/bin
1
以下以Relax程序作为举例:
# 首先进入工作目录,以Rosetta demo教程为例:
cd $ROSETTA/main/demos/tutorials/Relax_Tutorial
1
2
2
# 运行relax app relax.mpi.linuxgccrelease -s 1ubq.pdb -
relax:constrain_relax_to_start_coords -
relax:coord_constrain_sidechains -relax:ramp_constraints false -
ex1 -ex2 -use_input_sc -flip_HNQ -no_optH false
@general_relax_flags
1
2
3
4
5
2
3
4
5
输出结果如下